Técnicas de laboratório para identificação de Fusarium oxysporum f. sp. cubense |
|||||||||||||||||||||||||||||||||||||||||||
|
No laboratório, as amostras entregues para análise são conservadas no frio, a 8ºC, até à realização das análises. Antes de se iniciar as análises, é verificado se os sacos com as amostras estão corretamente identificados, com a referências de colheita e, de seguida, verifica-se o estado das amostras. Amostras mal-acondicionadas (não envolvidas em papel de filtro, desidratadas, ...), não referenciadas corretamente ou sacos com diferentes órgãos de planta são consideradas não conformes e rejeitadas.
As amostras conformes são registadas (Fig. 1). Retira-se o papel de filtro que as envolve, corta-se uma porção de tecido com uma faca desinfetada e esta porção é envolvida em papel de filtro. O material, depois de devidamente acondicionado, é transferido para uma câmara de fluxo laminar, onde existem condições de assepsia As técnicas que se aplicam às amostras de bananeira para identificação de Foc são: isolamento em meio de cultura, macroscopia, repicagem, microscopia e testes moleculares. No isolamento em meio de cultura, cortam-se pequenas porções de órgão da planta com zona de transição (Fig. 2A) e estas são transferidas para placas de Petri com meio de cultura Potato Dextrose Agar suplementado com o antibiótico estreptomicina (PDAs) (Fig. 2B). Posteriormente, as placas são seladas e incubadas de 25ºC durante 8 dias.
Os isolamentos são observados periodicamente, para se verificar o crescimento do fungo. Estabelecidos os crescimentos, as diferentes colónias obtidas são purificadas por repicagem. Retiram-se pequenas porções de micélio e estas são transferidas para placas de Petri com meio de cultura PDAs. Posteriormente, são seladas, incubadas a 25ºC e observadas periodicamente. Aos crescimentos obtidos, quer nos isolamentos quer nas repicagens, é aplicada uma técnica de macroscopia, ou seja, estes são observados visualmente e/ou à lupa binocular e caracterizados segundo alguns parâmetros, como diâmetro, coloração, presença/ausência de exudados e pigmentação (Fig 3A).
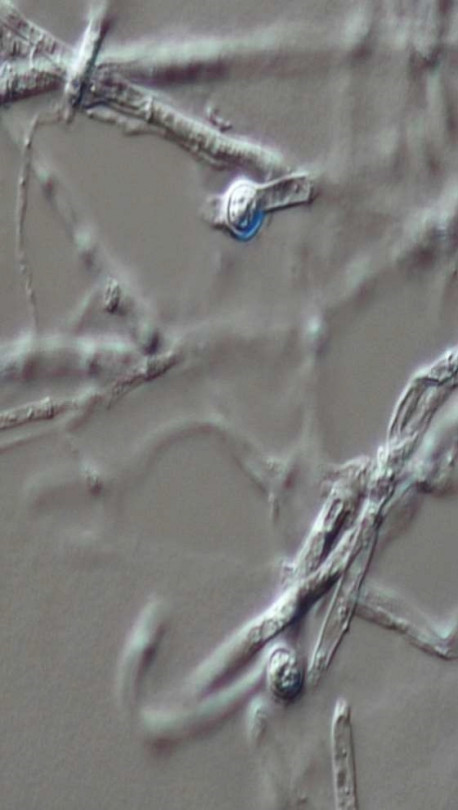
Como os fungos são de reduzidas dimensões, para a sua identificação realizam-se preparações microscópicas, para observá-los ao microscópio ótico. Estas preparação são importantes, pois na sua execução utiliza-se um corante, o qual cora as estruturas dos fungos, permitindo a identificação dos mesmos (Fig. 3B a 3D). Além das técnicas de laboratório que se consideram clássicas, também são aplicados outros testes para a identificação de Foc, nomeadamente testes moleculares. Estes são técnicas mais recentes, onde é utilizado para diagnóstico o ácido desoxirribonucleico (ADN) do fungo, sendo a Reação em Cadeia da Polimerase (PCR) a mais utilizada, na qual o ADN do fungo é amplificado a partir de amostras de plantas infetadas. Para a realização da PCR, é necessária efetuar o extrato do material que irá ser submetido a esta análise. Este material pode ser vegetal, (rizoma, pseudocaule etc.) ou micélio do fungo. |
Para o extrato de material vegetal, cortam-se pequenas porções dos órgãos da planta com sintomas (Fig. 2A), enquanto para extrato de micélio do fungo a partir dos crescimentos estabelecidos nas placas de Petri com meio de cultura PDAs, com uma ansa, retira-se micélio do fungo (Fig. 4). Ambos os extratos são colocados em tubos eppendorf (Fig. 5). para serem submetidos à extração de ADN (Fig. 6). Para o sucesso das técnicas posteriores, PCR convencional ou PCR em tempo real (qPCR), o ADN obtido, deve ser em quantidade e qualidade apropriadas, e uma vez obtido, pode ser conservado a 4 ºC por alguns dias, ou a -20 ºC por mais tempo, até posterior utilização. A PCR convencional é uma técnica que envolve a realização de uma mistura de reação (Mix), onde, num tubo de eppedendorf, são adicionados iniciadores específicos (primers) do fungo em análise e outros reagentes. Concluída a preparação da Mix, é distribuída em microtubos eppendorf e adiciona-se o extrato de ADN obtido das diferentes amostras, o controlo negativo e o controlo positivo. Posteriormente, os microtubos são transferidos para um termociclador pré-programado de acordo com o protocolo para a amplificação do ADN. Uma vez os produtos amplificados, estes são colocados em gel de agarose numa tina de eletroforese e, a seguir, procede-se à revelação em gel red. Quando o gel está corado, este é colocado num transiluminador com luz ultravioleta para leitura dos produtos amplificados da PCR, e regista-se os mesmos através de fotografia (Fig. 7).
Outro teste molecular que se pode aplicar para a identificação de Foc é o PCR em tempo real ou qPCR. É uma técnica molecular que permite monitorizar a amplificação do ADN em tempo real, ou seja, enquanto a mesma ocorre. Mais uma vez a partir dos extratos obtidos, com uma micropipeta é depositada numa microplaca ou em microtubos a mistura de reação (fornecida em kit próprio de diagnóstico) e o extrato de ADN das diferentes amostras, assim como, controlos negativos e positivos. Depois, a microplaca ou microtubos são colocados num termociclador de qPCR previamente programado com os ciclos, temperaturas e tempo, de acordo com o kit de diagnóstico (Fig. 8).
Decorridos alguns ciclos, no programa do computador ligado ao termociclador surgem diferentes curvas, uma por cada amostra e controlos positivos. Estas curvas indicam que o ADN está a ser multiplicado e a emitir fluorescência. Para o caso do(s) controlos negativos, como não existe ADN, este não se multiplica, logo não emite fluorescência, e em vez de uma curva, surge uma linha reta (Fig. 9).
Concluída a amplificação, é necessário interpretar os resultados obtidos, analisando as curvas obtidas e verificar se estas estão dentro dos valores referidos no kit de diagnóstico, e determinar se determinada amostra é ou não positiva. Existem mais técnicas moleculares descritas na bibliografia da especialidade para a identificação de Foc, no entanto, no presente artigo apenas descrevemos aquelas que o Laboratório de Qualidade Agrícola, na Camacha, integrado na Direção de Serviços dos Laboratórios Agrícolas e Agroalimentares, tem valências para as realizar, Bibliografia
|
||||||||||||||||||||||||||||||||||||||||||
- Detalhes